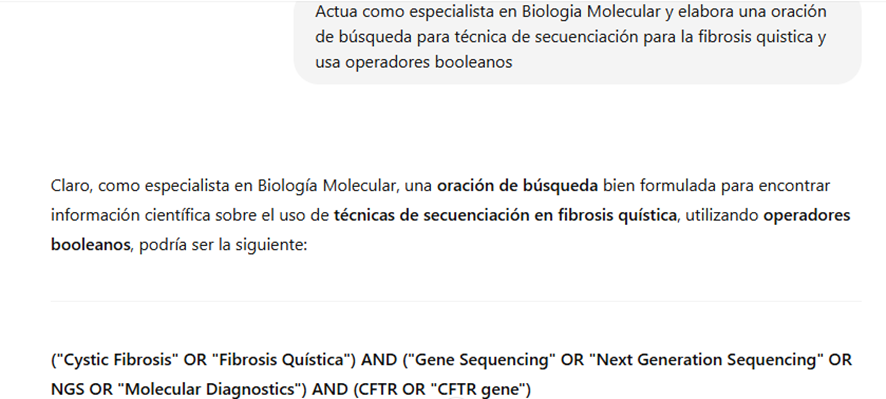
Técnica: RT-PCR (Reacción en cadena de la polimerasa con transcriptasa inversa)
Diagnóstico molecular está basado en la conversión del ARN viral en ADNc y su posterior amplificación usando sondas específicas (hibridación y amplificación basada en secuencia). Se utilizan genes diana como N, E, RdRp y ORF1ab para asegurar alta sensibilidad y especificidad. Esta técnica permite detectar bajas cargas virales. Las mutaciones del virus pueden afectar la eficacia de las sondas, lo que requiere actualizaciones continuas. Se usan alternativas como RT-LAMP y PCR digital.
Imagen obtenida de: http://www.scielo.org.co/scielo.php?script=sci_arttext&pid=S1794-24702020000300011
Referencias Bibliográficas
- Dhar. Diagnostic assay and technology advancement for detecting SARS-COV-2 infections causing the COVID-19 pandemic. PubMed Central [Internet]. 2022;414(9):2903-34. Disponible en: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC8872642/
- López BOP, Orozco BR, León JJC. SARS-CoV-2: generalidades bioquímicas y métodos de diagnóstico. Nova [Internet]. 25 de septiembre de 2020;18(35):11-33. Disponible en: http://www.scielo.org.co/scielo.php?script=sci_arttext&pid=S1794-24702020000300011